de l'infiniment petit
Nos équipes en bref
L’équipe Virologie Moléculaire & Cellulaire (VMC) se consacre à l’étude des virus à ARN brin positif. Les principaux objectifs de l’équipe sont de comprendre comment ces virus interagissent avec leur hôte à l’échelle cellulaire et moléculaire. Les modèles viraux étudiés sont le virus de l’hépatite C (HCV), le virus de l’hépatite E (HEV) et les coronavirus. Pendant plus de 28 ans, l’équipe a étudié le cycle infectieux du virus HCV et a largement contribué à la compréhension de plusieurs étapes de l’entrée, de la réplication et de l’assemblage de ce virus. En 2013-2014, après le développement d’antiviraux puissants contre le virus HCV, nous avons étendu nos modèles d’études au virus HEV et aux coronavirus hautement pathogènes tels que le MERS-CoV et depuis 2020 le SARS-CoV-2. Ces virus sont émergents dans les populations humaines et représentent une menace pour la santé. Notre objectif est de mieux comprendre leur cycle infectieux et d’identifier de nouveaux antiviraux.
Jean Dubuisson : Chef d'équipe
Pour en savoir plus sur l'équipe : ICI
Molecular and Cellular Aspects of the Hepatitis E Virus lifecycle : Laurence COCQUEREL
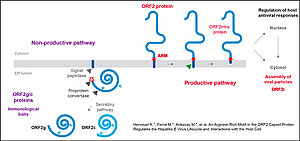
Le virus de l'hépatite E (HEV) est la principale cause d'hépatite virale aiguë dans le monde. Bien que l'infection par le HEV soit généralement spontanément résolutive, elle peut entraîner jusqu'à 30% de mortalité chez les femmes enceintes et conduire à une infection chronique chez les patients immunodéprimés. Il n'existe pas de traitement spécifique ni de vaccin universel pour lutter contre ce virus. Le HEV est un virus à ARN positif quasi-enveloppé exprimant trois cadres de lecture ouverts (ORF) : ORF1 qui code la réplicase virale ORF1, ORF2 qui code la protéine de capside virale ORF2 et ORF3 qui code une petite protéine impliquée dans la morphogenèse et la sécrétion des virions. En raison de la difficulté à amplifier le HEV en culture cellulaire, de nombreuses données manquent sur les différentes étapes du cycle infectieux du HEV.
Il y a quelques années, nous avons développé un système de culture cellulaire efficace qui permet d'étudier les mécanismes moléculaires et cellulaires du cycle infectieux du HEV. Ce modèle a notamment permis de démontrer de façon pionnière qu'au cours de son cycle infectieux, le HEV produit au moins 3 formes de la protéine de capside ORF2 : l'ORF2 infectieuse (ORF2i), l'ORF2 glycosylée (ORF2g) et l'ORF2 clivée (ORF2c). Ces formes ORF2 remplissent des fonctions distinctes dans le cycle infectieux du HEV. La protéine ORF2i est le constituant structurel des particules infectieuses tandis que les protéines ORF2g/c ne sont pas associées à du matériel infectieux mais agissent probablement comme un leurre de l'immunité humorale qui inhibe la neutralisation médiée par les anticorps. Les protéines ORF2g/c sont sécrétées en grandes quantités dans le surnageant de culture et sont les antigènes les plus abondants détectés dans le sérum des patients.
Très récemment, nous avons décrypté les mécanismes moléculaires de la multifonctionnalité de l'ORF2. En effet, nous avons identifié un motif riche en arginine (ARM), situé dans la région N-terminale de l'ORF2, qui contrôle la localisation subcellulaire, le devenir et les fonctions des formes ORF2. Il favorise également les interactions entre l'ORF2 et la cellule hôte. Nos observations ont mis en évidence que l'ARM de l'ORF2 est un régulateur central unique de l'adressage de l'ORF2 qui contrôle finement le cycle infectieux du HEV.
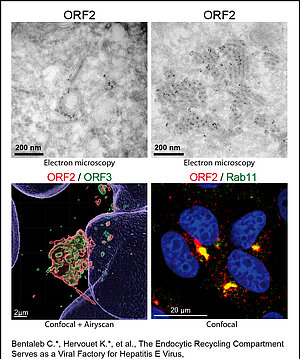
Dans une autre étude, nous avons généré des anticorps monoclonaux qui reconnaissent spécifiquement la forme ORF2i associée aux particules virales, et des anticorps qui reconnaissent les différentes isoformes de l'ORF2. Nous les avons utilisés dans des approches de microscopie confocale et électronique pour sonder les particules infectieuses et les usines virales dans les cellules produisant le HEV. Nous avons identifié un réseau membranaire sans précédent induit par le HEV, contenant des structures tubulaires et vésiculaires qui sont enrichies en protéines virales ORF2 et ORF3. De plus, des analyses approfondies de colocalisation des protéines virales avec des marqueurs cellulaires ont démontré que ces structures sont dérivées du compartiment endosomal de recyclage (ERC)
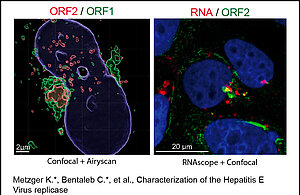
De plus, dans une étude visant à caractériser la réplicase ORF1 et utilisant la technologie RNAscope, nous avons révélé que la protéine ORF1 et l'ARN génomique sont également enrichis dans les structures subcellulaires dérivées de l'ERC (Illustration n°3), indiquant que l'ERC sert d'usine virale pour le HEV. Par conséquent, nos études ont démontré que le HEV détourne l'ERC pendant son cycle infectieux et forme un réseau membranaire de structures vésiculaires et tubulaires qui pourrait être la marque de l'infection par le HEV.
Morphogenèse des coronavirus : Sandrine BELOUZARD
La nucléocapside des coronavirus est protégée par une enveloppe lipidique dans laquelle sont enchâssées trois protéines virales. La protéine spike (S) forme des protrusions à la surface des virus donnant un aspect caractéristique à cette famille de virus en de microscopie électronique. Cette protéine est responsable de l’entrée du virus dans la cellule cible ce qui en fait la principale responsable du tropisme cellulaire. La protéine de membrane (M) est la protéine la plus abondante de l’enveloppe virale, avec la petite protéine d’enveloppe (E), elle joue un rôle important pour l’assemblage des nouvelles particules virales.
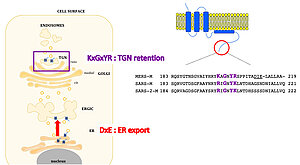
La protéine M est composée d’un court domaine N-terminal glycosylé suivi de 3 passages transmembranaires et d’un long domaine C-terminal qui représente environ la moitié de la protéine. Des interactions de la protéine M avec toutes les autres protéines structurales ont été démontrées (E, N et S), c’est pourquoi elle est considéré comme l’élément moteur de l’assemblage viral. L’assemblage des coronavirus est réalisé dans le compartiment intermédiaire entre le Reticulum Endoplasmique (RE) et le Golgi (ERGIC). Cette formation de nouvelles particules virales a deux pré-requis (1) tous les éléments doivent être adressées au compartiment de bourgeonnement des virus et (2) une série d’interactions doit être mise en jeu non seulement entre les composants structuraux (M-M ;M-E ;M-N et M-S) mais probablement avec des facteurs cellulaires afin d’assurer l’incorporation correcte des protéines structurales et la sécrétion des virus. Le but de ce projet est de mieux comprendre les mécanismes d’assemblage des coronavirus en se focalisant d’abord sur la protéine M. Plus précisément, notre objectif est d’identifier des régions de la protéine M importante pour l’assemblage des virus et de caractériser leur mécanisme d’action. Lorsqu’elles sont exprimées seules, les protéines M des coronavirus sont localisées dans le Golgi, pour les protéines MERS-M et SARS-2-M elles sont localisées dans le réseau trans-Golgien (TGN). Dans la protéine M du MERS-CoV, nous avons identifié deux signaux importants pour son trafic intracellulaire : un signal DxE pour son export du RE et un signal KxGxYR qui participa à sa rétention dans le TGN. La mutation de ce dernier entraîne l’expression de la protéine à la surface des cellules et inhibe la formation de nouvelles particules virales. Le motif KxGxYR est très conservé chez les betacoronavirus dont le SARS-CoV-2. Actuellement, nous étudions le trafic intracellulaire de la protéine SARS-2-M. A plus long terme, nous souhaitons aussi identifier des facteurs cellulaires impliqués dans la morphogenèse virale et comment la protéine M pourrait les recruter.
Composants Naturels compounds & Antiviraux : Karin SERON
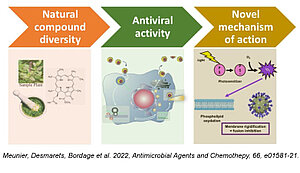
L'identification d'antiviraux ciblant différentes étapes du cycle viral peut servir de base à la découverte de nouvelles thérapies et peut aider à mieux comprendre le cycle infectieux viral. Les plantes sont utilisées depuis des siècles en médecine traditionnelle pour soigner différents types de maladies, et les produits naturels d’origine végétale sont à l’origine de la découverte de nombreux médicaments. Même si ces produits naturels ne sont pas toujours utilisés directement comme médicaments, ils restent souvent une source d'inspiration pour la chimie médicinale. Récemment, nous avons concentré nos recherches sur les antiviraux d'origine végétale contre le SARS-CoV-2. Nous avons criblé des extraits de plantes pour leur activité antivirale contre les coronavirus et identifié un extrait de plante de Côte d'Ivoire (sélectionné par Moussa Bamba, doctorant et Pr. Fézan Tra Bi, Université Nangui Abrogoua, Abidjan) à très haute capacité antivirale. Grâce à une collaboration avec le laboratoire de pharmacognosie de la Faculté de Pharmacie de Lille (Pr Sevser Sahpaz et Dr Simon Bordage), l'extrait a été fractionné selon une procédure de fractionnement bio-guidée et nous avons pu identifier le composé actif, le « phéophorbide a ». Ce composé est un produit de dégradation de la chlorophylle et un photosensibilisateur. Nous avons montré que l'activité antivirale du phéophorbide est dépendante de l'activation lumineuse et induit une rigidification de l'enveloppe virale. Ceci a été observé par cryo-microscopie électronique (collaboration avec Olivier Lambert et Marion Decossas, CBMN UMR 5248, Bordeaux). Le phéophorbide est le premier antiviral naturel décrit contre le SARS-CoV-2 avec une activité virucide activable par la lumière et qui présente un potentiel pour la thérapie anti-COVID-19 ou la désinfection des surfaces contaminées par le SRAS-CoV-2. Nous avons également montré que le phéophorbide était capable d’inactiver d’autres virus tels que le virus de l’hépatite C et le virus de la fièvre jaune. De nouveaux projets sont en cours pour explorer les capacités antivirales d’autres molécules d'origine végétale.
Mécanismes moléculaires et cellulaires à l'origine des altérations des peroxysomes dans les cellules infectées par le virus de l'hépatite C : Yves ROUILLE
Le virus de l'hépatite C (VHC) est un important agent pathogène humain qui infecte le foie et établit fréquemment une infection chronique, entraînant à plus long terme des complications telles que stéatose, fibrose, cirrhose et carcinome hépatocellulaire. Malgré les progrès récents du traitement de l’hépatite C, le risque de complications induites par le VHC est réduit, mais pas complètement éliminé chez les patients qui ont résolu leur infection.
Les mécanismes moléculaires et cellulaires conduisant aux troubles pathologiques sont encore mal compris. Au niveau cellulaire, l'infection par le VHC induit des altérations morphologiques et métaboliques. Un certain nombre de facteurs cellulaires sont recrutés par le virus pour réaliser les différentes étapes de son cycle de vie. Ce recrutement peut détourner certains facteurs cellulaires recrutés de leur localisation et de leur fonction intracellulaires normales, ce qui peut interférer avec le métabolisme cellulaire et contribuer ainsi à la pathogenèse.
Un projet visant à identifier les facteurs des cellules hôtes recrutés au niveau des sites de réplication du VHC a conduit à l'identification d'ACBD5 (Acyl-CoA-binding domain-containing protein 5). ACBD5 est une protéine membranaire des peroxysomes qui impliquée dans la formation des sites de contact membranaire entre peroxysomes et réticulum endoplasmique (RE), via une interaction avec VAP-A/B, deux protéines de la membrane du RE qui sont importantes pour la réplication du VHC. Des expériences de microscopie confocale ont confirmé le recrutement des peroxysomes à proximité des complexes de réplication du VHC et ont également révélé des altérations de leur morphologie. Nous étudions l'interaction entre le VHC et les peroxysomes en évaluant la fonction des peroxysomes dans la réplication du VHC et en analysant l'impact de l'infection par le VHC sur la fonction des peroxysomes, avec un focus particulier sur le stress oxydatif. Nous attendons de ce projet qu’il révèle des mécanismes moléculaires et cellulaires induits par le VHC pour altérer le métabolisme cellulaire et plus particulièrement les fonctions des peroxysomes, dont on sait qu'elles déclinent dans les hépatocytes des patients infectés de façon chronique.
Team « Research on Mycobacteria and Bordetella »
Our projects are focused on two major bacterial respiratory pathogens: Mycobacteria and Bordetella.
Today, respiratory infections, many of which are caused by pathogenic bacteria, are among the world’s most successful killers.
Tuberculosis (TB) is the cause of approximately 1.8 million annual deaths. Pertussis, the existence of which had almost been forgotten, shows a dramatic global re-emergence, including in high-vaccine coverage countries.
The choice of working in parallel on these two bacterial models is motivated by the fact that whereas both bacteria infect the human respiratory tract, they follow extremely different strategies that we believe are interesting to compare. For both infections, vaccines are available but the re-emergence of both diseases indicates that there is a need for improvement. Moreover, while antibiotic resistance of B. pertussis is currently not a concern, the global spread of multidrug resistant TB is a major problem.
The objectives of the team are :
(i) to study the molecular mechanisms of pathogenicity of B. pertussis and M. tuberculosis pathogen families,
(ii) to analyse the genome evolution of M. tuberculosis and the genetic regulation of the two pathogens,
(iii) to use this knowledge to develop novel approaches to design better vaccines, new therapeutics and new methods for molecular diagnostics and molecular surveillance, which are urgently needed to fight against these deadly scourges.
Thus, our research is a continuum from a continuous acquisition of basic knowledge to the development of clinical applications and investigations. These ambitious objectives are led with the help of collaborations with many academic French collaborators, including within the CIIL, and international laboratories, in the framework of multiple ANR and EU programmes, and with very active private partners participating to the clinical and industrial development of our discoveries.
Main projects of the team RMB
Molecular mechanisms of pathogenicity of B. pertussis and M. tuberculosis pathogen families
Handling of copper by a host-restricted pathogen.
A recent project in the laboratory aims at elucidating the homeostasis of copper, an important player at the host-pathogen interface, in Bordetella pertussis. As a strict aerobe, B. pertussis needs copper notably for its heme-copper cytochrome oxidases. However, copper is also toxic and is notably used by phagocytic cells to kill bacteria. We have discovered that B. pertussis has shed most of its defenses against copper and conversely has acquired a specific copper acquisition system which is tightly regulated. It also produces a new type of natural product that binds copper and most likely participates in its homeostasis. (contact: francoise.jacobibl.cnrsfr)
Mechanisms of intracellular degradation of B. pertussis
Xenophagy represents a highly conserved defense mechanism of eukaryotic cells involved in the clearance of invading pathogens. During this complex process, intracellular micro-organisms are targeted to the degradative lysosomal compartment. A non-canonical autophagic pathway through LC3-associated phagocytosis (LAP) also contributes to immune regulation and inflammatory responses.
Once therespiratory pathogen B. pertussis is inhaled, it will encounter phagocytic cells which represent a first line of defense against the infection. Among them, alveolar macrophages play a key role in protection. A knowledge gap exists on the involvement of autophagy and/or LAP in the intracellular clearance of B. pertussis and its impact on innate immune responses. We established a novel partnership with Dr. Ghaffar Muharram (MCPI team, CIIL) to tackle this question. (contact: nathalie.mielcarekinsermfr; carine.rouanet@ibl.cnrs.fr)
Implication of protein-protein interactions in mycobacterial pathogenesis
Deciphering the Protein-Protein Interactions (PPI) in pathogenic bacteria may help to understand the physiology of the cell and to elucidate host-pathogen interactions, in which proteins play crucial roles. In addition, the study of PPI may facilitate the discovery of protein functions by the ‘guilty by association’ principle.
As PPI are key factors in Mycobacterium tuberculosis physiology and virulence, we are particularly interested in deciphering PPI network in mycobacteria at the cell wall and the membrane level. For example, mycolic acid biosynthesis, which is the target several antiTB drugs, relies on specialized and interconnected protein complexes. Hence, the identification and the characterization of PPI may represent an attractive approach for the development of new drugs and/or peptidomimetics. In addition, deciphering the PPI network of M. tuberculosis will lead to the identification of critical steps required for mycobacterial infection and will allow a better understanding of TB pathogenesis. (contact: romain.veyron-churletibl.cnrsfr)
Genetic regulation and genome evolution
Bordetellae’s gene expression regulation
The Bordetellae, encompassing the human pathogen B. pertussis and the veterinary pathogen B. bronchiseptica, produce an arsenal of virulence factors such as adhesins and toxins, that allow the bacteria to transmit, infect and colonize the respiratory tract of the host. These bacteria regulate their virulence factor expressions according to environmental conditions. Hence, these species have a complementary set of capabilities that is regulated in an inverse fashion. In the virulence phase the virulence-activated genes or vags are transcribed and their expression is regulated by a two-component system BvgAS. In the a-virulence phase, the vags are not expressed but instead, the virulence-repressed genes or vrgs are expressed. The expression of the vrgs is regulated by another two-component system RisA/K and by the action of BvgR, a c-di-GMP phosphodiesterase. Using molecular biology and omics’s technologies, our goal is to define the regulation network and the mode of regulation involving the BvgASR and the RisAK systems. Additionally, B. pertussis encodes for up to 16 different two-component systems arguing on the extreme complexity of the Bordetella gene expression biology. (contact: loic.coutteinsermfr)
Evolutionary history and factors driving the spread of tuberculosis

Mycobacterium tuberculosis is the deadliest bacterial infectious agent globally and the first contributor to antimicrobial mortality. Using comparative genomics and pathophysiological approaches applied to strain lineages with different epidemic and/or antibiotic resistance profiles, we aim at identifying the factors that have contributed to its emergence and its exceptional evolutionary success, including in multidrug-resistant (MDR) forms.
This work has led to the discovery of exceptional ancestral branches of tubercle bacilli in East Africa, which was the foundation for the identification of novel molecular mechanisms of virulence/persistence of the pathogen in the host. We also used/use whole genome sequencing to reveal the longitudinal spread of epidemic MDR clones, and to expand knowledge on drug resistance associated mutations, which is necessary for maximizing the accuracy of novel molecular diagnostic tools. (contact: philip.supplyibl.cnrsfr)
Translational research
BPZE1 vaccine against pertussis

Decades of research on the molecular pathogenesis of pertussis have allowed us to develop a novel, live attenuated nasal pertussis vaccine called BPZE1. In contrast to the currently available vaccines, this vaccine protects both against pertussis disease and infection by the causative agent Bordetella pertussis, as assessed in mice and non-human primates. It is now in clinical development and has successfully completed two phases I and two phases II clinical trials. These trials have shown that the vaccine is safe in humans, able to transiently colonize the human respiratory tract, to induce both humoral and T cell responses and to prevent subsequent colonization by a second dose of the vaccine strain. In addition to continuing its clinical development, current research focuses on using the vaccine strain for the presentation of heterologous antigens to the respiratory mucosal immune system. (contact: camille.lochtinsermfr; nathalie.mielcarekinsermfr)
Deep sequencing for culture-free diagnosis of drug resistance in mycobacteria (Deeplex Myc-TB and Deeplex Myc-Lep)
Less than 40% of the estimated 460,000 new TB cases with rifampicin resistance or multidrug-resistance occurring each year are diagnosed and treated, reflecting important limitations of conventional phenotypic and molecular tests. By building on the impressive progress of next-generation sequencing (NGS) technologies and the knowledge gained on the catalogue of resistance determinants in the M. tuberculosis genome, novel tools for rapid NGS-based, culture-free diagnostics are developed by ©GenoScreen (Lille) with our collaboration. This has led to the development of the Deeplex® Myc-TB kit, based on deep amplicon sequencing for prediction of susceptibility or resistance to 13 anti-tuberculosis drugs/drug classes, directly applicable on clinical samples. As a unique feature compared to other commercial molecular tests, this assay also includes genotypic detection of recently re-defined extensively drug resistant (XDR) tuberculosis. It is used by the WHO for surveillance of drug resistant TB and in >25 countries, and is being evaluated in several MDR-TB diagnostic trials, e.g. in Africa in the DIAMA H2020 EDCTP project. A similar test is being developed for culture-free diagnosis of the agent of leprosy, M. leprae, which is unculturable in vitro. (contact: philip.supplyibl.cnrsfr)
INFLAMMAVAX (Anti-inflammatory benefits of BPZE1)

With advancing age, the immune system undergoes a dynamic change characterized by the coexistence of a weaker immune response to newly encountered pathogens or vaccine antigens, and a systemic inflammatory state that is involved in the development of cardiovascular disease, metabolic disorders and cancer in the elderly. During the development of the attenuated live pertussis vaccine (BPZE1) in our laboratory, we have observed non-specific anti-inflammatory properties that protect against morbidity and mortality associated with inflammation induced by heterologous viral or bacterial infections, as well as against non-infectious inflammatory disorders, such as allergic asthma. In our project, we plan to understand the molecular mechanisms behind these protective anti-inflammatory effects. The basic knowledge resulting from our project will open new avenues for the prevention of chronic inflammation, the common denominator of all age-related diseases. In the long term, our study may thus lead to novel anti-inflammatory therapies promoting healthy longevity. (contact : stephane.cauchiibl.cnrsfr)
Small Molecules Aborting Resistance against Tuberculosis (SMARt-TB)

Alarmingly, multidrug-resistant and extensively drug-resistant Mycobacterium tuberculosis have now spread worldwide. Some key antituberculosis antibiotics are prodrugs, for which resistance mechanisms are mainly driven by mutations in the bacterial enzymatic pathway required for their bioactivation. We are developing molecules that stimulates alternative prodrug activation pathway called Small Molecules Aborting Resistance (SMARt).
SMARt molecules aborting ethionamide resistance have been discovered in collaboration with team U1177 (ANR-IPL-Région HdF). First-in human study was initiated by Bioversys and GSK in 2020 with a molecule reverting Ethionamide resistance. A second family of compound aborting Pretomanid and Delamanid resistance is under evaluation with the support of ANR and SATT-Nord. The bioactivation of fluoroquinolone derivatives is also studied in collaboration with the team of Alexandra Aubry (Paris-CIMI), supported by the ANR project Detonator. (Contact: alain.baulardinsermfr )
Our team coordinates the “Mustart” project recently funded by the French initiative “Investments for the Future”(PIA3). Mustart associates 9 French academic experts to develop multifaceted regimens targeting the most coriaceous TB bacilli in their peculiar physiological niches, and to identify biomarkers of the treatment progress. (Contact: alain.baulardinsermfr )
Témoignages
Alix Laffitte, doctorante, et Servane Le Guillouzer, Post-doctorante, dans l’équipe Peste et Yersinia pestis, retracent leurs parcours académiques et leurs plans pour le futur. Elles vous présenteront leurs thématiques de recherche. Découvrez leurs témoignages!
La peste a été éradiquée? ... Par Florent SEBBANE, responsable de l'équipe Peste & Yersinia Pestis au CIIL

Le dernier cas en France remonte à 1945, mais a-t-elle disparu ?
Depuis l’Antiquité, elle a tué 200 millions de personnes. Le traumatisme en Occident fut tel qu’il imprègne encore notre vocabulaire, que l’on qualifie des personnes (« une peste », « un pestiféré »), des odeurs (pestilentielles) ou un dilemme infernal (« Choisir entre la peste et le choléra »). « En France, après la peste des chiffonniers à Paris en 1920, on trouve un dernier cas en Corse en 1945 », relate Florent Sebbane, responsable du laboratoire Peste et Yersinia pestis de l’Inserm.
Pour en savoir plus : ICI
La peste, considéré comme une maladie du passé, n’a jamais été éradiqué. Dans ce reportage de l’émission 36.9 (Radio Télévision Suisse), découvrez les travaux menées par les équipes du Dr Florent Sebbane à l’Institut Pasteur de Lille et du Dr Javier Pizarro-Cerda à l’Institut Pasteur à Paris sur la peste: de la compréhension des mécanismes de transmission par les puces au développement de vaccin.

Pour plus de détails sur l'équipe --> ICI
Les infections par des protozoaires tels que Plasmodium, Leishmania, Toxoplasma provoquent plus de maladies asymptomatiques chroniques que de maladies sévères voire mortelles dans les zones de forte endémicité. Les individus asymptomatiques développent une immunité anti-maladie et deviennent des réservoirs de parasites. Dans les écosystèmes naturels, la diversité des populations de parasites, l'environnement partagé et/ou individuel, y compris la réponse immunitaire de l'hôte et le patrimoine génétique contribuent ensemble à l'établissement de l’immunité. Cependant, dans de tels contextes, l’immunité peut être influencée par des infections commensales et simultanées avec d’autres parasites constituant le «multi-biome» de l’hôte. L’équipe développe une stratégie globale et intégrée «one health» associant une recherche clinique et fonctionnelle, en capitalisant sur l'expertise pluridisciplinaire des membres de l’équipe (cliniciens travaillant sur le terrain, immunologie, parasitologie et épidémiologie) afin 1) d’identifier les signatures immunologiques associées aux formes asymptomatiques ou pathologiques des infections et 2) d’évaluer l’impact de leurs modulations par les facteurs environnementaux et le multi-biome de l’hôte. Les résultats obtenus devraient permettre de mettre en évidence des biomarqueurs phénotypiques et des cibles thérapeutiques pour le diagnostic, le pronostic, la prévention et le traitement.
L'équipe est répartie sur deux sites : 1) à l'Institut Pasteur de Lille dirigée par S. Pied DR CNRS et 2) à l'Université de Guyane et du Centre Hospitalier de Cayenne (CHC) dirigée par M. Demar, PU-PH. L'équipe est liée au Laboratoire de Parasitologie-Mycologie et à plusieurs unités médicales, dont les services de 1) maladies infectieuses et tropicales, 2) de dermatologie, et 3) plusieurs centres de santé pour les zones reculées de Guyane. TBIP a aussi des liens étroits avec le Centre d’Investigation Clinique (CIC INSERM-CIE 802 dirigé par M. Nacher).
A travers les épisodes hebdomadaires, vous découvrirez nos différentes thématiques de recherche lilloises et guyanaises. Pour cette première semaine, zoom sur la Toxoplasmose amazonienne…
Episode 1
Episode 2
Episode 3
Episode 4
Team Bacteria, Antibiotics, and Immunity
Respiratory tract infections represent the third major cause of death worldwide. Among them, bacterial pneumonia (either community- or hospital-acquired) are major causes of morbidity, quality-adjusted life year loss, and mortality in children, adults, and the elderly. Although the discovery of antibiotics to treat bacterial pneumonia was a remarkable achievement in the 20th century, the effectiveness of antibiotics is declining, because of antimicrobial resistance (AMR). The World Health Organization (WHO) estimates that bacterial infections due to AMR will outcompete any cause of death by 2050, meaning that it is crucial to develop new strategies to improve antibacterial treatment.
The team "Bacteria, Antibiotics, and Immunity" studies pathogenic bacteria responsible for pneumonia listed by the WHO as priority targets in the fight against AMR: Streptococcus pneumoniae and Klebsiella pneumoniae. Our studies make use of cell and animal models as well as clinical studies with the GHICL hospitals in Lille to translate findings from bench to bedside. The team especially investigates the immune responses that are triggered by the bacterial infection of the respiratory tract, by the antibiotics during the treatment of such infections and aims to define groundbreaking interventions that increase innate immune defenses. Notably, innate immunity mobilizeq a variety of antibacterial effectors; Emergence of resistance to these immune effectors would be unlikely. The ultimate objective of the team is to combat the emergence of AMR by novel interventions that combine stimulation of innate immune responses and antibiotherapy.
The team currently explore three research axes:
- Demonstrating the proof-of-concept of aerosol delivery of immune-modulator as an innovative pneumonia treatment
- Defining the contribution of innate immune effectors in the effectiveness of antibiotic treatment of infections
- Dissecting the mechanisms involved in the innate IL-17/IL-22 responses to develop immune-boosters of antibiotics
In conclusion, our studies tests and optimizes emerging concepts of immunotherapy of bacterial pneumonia in combination with the standard of care that are antibiotics in order to address the problem of AMR.

Demonstrate the proof of concept of aerosol delivery of immune-modulator as an innovative pneumonia treatment
The team coordinates the European project FAIR (https://fair-flagellin.eu) that aims at targeting the innate immune system as an underexploited area of drug discovery for infectious diseases. To this purpose, the FAIR project develops inhaled flagellin as an adjunct therapy in bacterial pneumonia. Flagellin is an agonist of the Toll-like receptor 5 that activates signaling in the respiratory epithelium and thus, protective innate defenses against pneumonia. It has already been shown that flagellin activates local or systemic innate immunity and enhances the therapeutic outcome of pneumonia caused by antibiotic susceptible or resistant S. pneumoniae and K. pneumoniae. We aim at dissecting the mode of action of flagellin during pneumonia treatment. The project also ambitions to setup a first-in-man phase I clinical trial.

Decipher the nature of innate immune effectors contributing to antibiotics efficacy during bacterial pneumonia
Antibiotics are uniquely considered as direct antimicrobial agents, and most efficacy evaluations are based on in vitro assays or immunosuppressed animals. Growing evidence suggests that antibioticsinteract with host innate immunity to provide potent indirect effects which enhance bacterial clearance and may result in more rapid and complete therapeutic effects. The team is characterizing the dynamics of myeloid cell differentiation and functions during the antibiotic treatment of bacterialinfection in mice, in order to identify novel immunomodulatory agents.

Dissect the innate IL-17/IL-22 responses to improve treatment of pneumonia and boost antibiotic effectiveness.
The innate lymphocytes that produce the cytokines IL-17 and IL-22 are essential to promote respiratory innate antimicrobial defenses and tissue repair. The team aims at understanding how these responses are induced in the respiratory tract during self-limiting bacterial pneumonia in order to develop innovative interventions.
Our new Post doc
Our new PhD students

Mara Baldry - Postdoctoral researcher Scientific manager of FAIR. Immune corelates of protection to inhaled flagellin as adjunct of antibiotics.

Charlotte Costa - PhD Student. Impact of immunotherapies on the treatment and emergence of antibiotic-resistant pneumonia.

Mélanie Mondemé - PhD Student. Dynamics of myeloid cells during antibiotherapy of pneumonia.

Xing Li - PhD Student. Innate response of human primary respiratory cells to infections and immune-modulators.

Yasmine Zeroual - PhD Student. Modeling of protective innate immunity by Omics analysis.
Pour plus de détails sur l'équipe : ICI
Les maladies respiratoires chroniques constituent une des causes majeures de mortalité et de morbidité. L’appareil respiratoire se situe à l’interface entre l’environnement et l’hôte, et joue un rôle majeur dans le développement de la réponse immune de l’hôte bénéfique ou délétère. Cette réponse est une des voies à cibler pour modifier l’évolution naturelle des maladies respiratoires. Parmi elles, notre équipe étudie deux pathologies respiratoires, le syndrome de détresse respiratoire aigu (SDRA), très souvent d’origine infectieuse et l’asthme sévère. Du fait de nos liens forts avec le service de pneumologie et de réanimation du CHU de Lille, nous sommes très focalisés sur le retour aux patients de nos travaux de recherche.
Dans le cadre du SDRA, un lien a été montré entre des taux bas d’endocan, un protéglycane endothélial cloné au laboratoire, et le développement d’un SDRA lors d’un choc septique (Gaudet et al, J Crit Care, 2018). Ceci nous a conduit à évaluer l’endocan dans une cohorte de patients atteints de COVID ayant séjourné dans le service de réanimation de l’hôpital Foch à Suresnes. Les résultats ont montré que l’endocan était plus élevé chez les patients COVID versus non COVID, et que les patients développant les SDRA les plus sévères avaient les taux les plus bas d’endocan entre J3 et J5 après l’entrée en réanimation, suggérant un rôle prédictif de cette molécule dans le développement d’un SDRA chez les patients COVID (Pascreau et al, Crit care, 2021).
Dans le cadre de l’asthme, celui-ci demeure un challenge majeur du fait de la multiplicité de ses phénotypes cliniques et de ses mécanismes biologiques. Si l’asthme sévère ne représente que 5 à 10% de la totalité des asthmes (qui touche quand même plus de 300 millions de personnes dans le monde), c’est cette forme qui génère la quasi totalité des dépenses de santé afférentes à cette pathologie. On distingue schématiquement les asthmes T2 (avec un profil éosinophilique) et les asthmes non-T2 (avec des profils neutrophiliques ou mixtes). Ces asthmes sont sujets à de fréquentes exacerbations, qui peuvent être provoquées par des infections, des expositions allergéniques, ou des agressions par les polluants. C’est dans ce contexte que l’équipe immunité pulmonaire, s’intéresse aux mécanismes immunobiologiques de l’asthme, afin de proposer de nouvelles cibles thérapeutiques, en décortiquant les mécanismes dans des modèles animaux, et en les évaluant dans des cohortes de patients asthmatiques.
Ainsi nous avons montré dans un modèle expérimental T2 d’asthme aux acariens, que la détection par NOD1 de certaines bactéries associées aux acariens, aggrave la sévérité de l’asthme in vivo, et qu’inhiber cette voie pourrait être une approche thérapeutique pour traiter l’asthme (Ait Yahia et al, J Allergy Clin Immunol, 2021). Dans un modèle cette fois ci non T2 neutrophilique induit par l’allergène de chien et générateur d’un fort remodelage bronchique, l’inhibition de voies conduisant à la production d’IL-22 atténue ce phénotype (Bouté et al, Allergy, 2021). Vous trouverez plus bas deux vidéos 3D montrant le remodelage bronchique par la visualisation par microscopie bi-photonique des fibres collagènes (en vert) soit au niveau du poumon en base, soit dans le modèle d‘asthme non T2 (collaboration E Weirkmeister, équipe microbiologie cellulaire et physique de l'infection). Comme il n’existe actuellement aucune biothérapie ciblant les asthmes non-T2, ces résultats sont très prometteurs et des études de « preuves de concept » sont actuellement en cours (START AIRR soutenu par la région Hauts de France)
Ainsi nos travaux mettent en exergue différentes voies qui pourraient constituer des biothérapies du futur dans l’asthme sévère.
les fibres de collagène de type I peri bronchiques sont visualisées en vert par la génération de signaux de seconde harmonique (SHG) en microscopie bi-photon au niveau du poumon en base. Les autres structures du poumon apparaissent en rouge.
les fibres de collagène de type I peri bronchiques sont visualisées en vert par la génération de signaux de seconde harmonique (SHG) en microscopie bi-photon au niveau du poumon après induction d’un asthme allergique au chien. Notez les fibres en triple hélice du collagène.

Pour plus de détails sur l'équipe CMPI : ICI

Notre équipe étudie les interactions entre les cellules et les tissus d’une part et, d’autre part, les bactéries, toxines, peptides antimicrobiens, virus en s’intéressant à la réponse mécanique et biologique suscitée. Lors de l’interaction, mécaniquement, des modifications structurelles sont induites, qui en retour vont moduler une réponse cellulaire, voire tissulaire.
La réponse que nous étudions est liée au mécanisme de l’autophagie qui permet notamment à la cellule de dégrader des microbes qui rentrent dans les cellules, mais certains de ces microbes ont développé, au cours de l’évolution, des systèmes pour y échapper voire l’exploiter.
Suivant les propriétés mécaniques des cellules, structurellement, la fluidité, l élasticité, la viscosité sont modifiées. Ceci concerne des molécules, membranes et compartiments intracellulaires. L’ensemble participe à définir la capacité de réponse à l’interaction micro-organisme/cellule ou tissu.
C’est pourquoi, nous développons une approche multidisciplinaire alliant biologie et physique et concevons des méthodes innovantes qui nous permettent de lever des verrous conceptuels en ce qui concerne ces interactions entre hôte et microbes. Si l’on peut contrôler ces interactions ou les réponses qu’elles entrainent alors nous pouvons moduler leurs conséquences.
Nous vous proposons 4 vidéos courtes qui après une rapide introduction sur la problématique générale, décrivent des sujets actuellement étudiés dans le Groupe :
1- Mécanobiologie de la molécule à la cellule et aux tissus
2- Technologies innovantes en interactions hôte-pathogènes
3- Les effets bénéfiques pour la santé des bactéries lactiques dans l’intestin
4- Les réponses cellulaires aux infections à norovirus.
Bon visionnage !
Subjet #4
Subjet #3
Subjet #2
Subjet #1
La (multi)résistance aux antibiotiques est un problème de santé mondial important et croissant, notamment pour la tuberculose et de nombreuses infections bactériennes nosocomiales. À l'appui de ce constat, l'Organisation Mondiale de la Santé (OMS) a proclamé que la recherche et le développement de médicaments pour combattre ces bactéries résistantes aux médicaments étaient une priorité absolue. L'équipe de BCA (Biologie Chimique des Antibiotiques) étudie une série de nouvelles stratégies visant à combattre les bactéries résistantes aux antibiotiques. Les recherches actuelles se concentrent sur la découverte et le développement de nouveaux antibiotiques et sur le développement de stratégies innovantes qui améliorent la pénétration des antibiotiques dans les bactéries.

Pour plus de détails sur l'équipe CBA : ICI
Les travaux de l'équipe Grippe, Immunité & Métabolisme (I2M) ont pour objectif de mieux comprendre les conséquences physiopathologiques des infections respiratoires virales. Nous nous intéressons à la grippe et au SARS-CoV-2, l’agent de la COVID-19. Nous étudions les événements se déroulant dans les poumons, mais également en dehors des poumons. Dans notre démarche, nous intégrons l’impact de l’âge et des comorbidités (dyslipidémie) dans la réponse de l’hôte lors de l’infection. Cette recherche intégrée nous a récemment conduits à mieux appréhender l’influence de l’axe intestin-poumon et particulièrement à identifier le microbiote intestinal comme un acteur de la pathologie liée à l’infection. Le tissu adipeux est également sollicité au cours de l’infection et nous tentons d’en comprendre les conséquences. Pour mener à bien ses recherches, l’équipe I2M développe des modèles expérimentaux en lien avec les cliniciens et tente de proposer des solutions thérapeutiques qui pourraient être exploitées chez l’homme.
Pour en savoir plus sur l'équipe: ICI
Equipe OPINFIELD
L'une de nos préoccupations majeures concerne la 3ème cause de décès dans le monde: la COPD.
Vous souhaitez en savoir plus ainsi que son application thérapeutique? Suivez notre série de 3 épisodes.
Episode 3
Episode 2
Episode 1